【学术前沿】特别致敬CRISPR幕后的英雄们——最全的一份CRISPR英雄谱
以下文章来源于BioArt ,作者小鹿

BioArt
高屋建瓴,提供专家点评,引导学术争论,展现学术批评;诚心实意,关注科研生态,推广科研经验,倡导师生交流。

关注我们,获取更多相关资讯

BioArt按:Jennifer Doudna和Emmanuelle Charpentier获得诺奖,被很多人认为实至名归。然而重大的科学发现经常是站在巨人肩上获得的,在CRISPR基因编辑技术发现史上,还有很多英雄,虽然他们没有获得诺奖,但是他们的贡献是伟大的。关于CRISPR基因编辑技术发现史已有无数的文章过去报道过,但是最有影响的还是2016年1月14日,Cell杂志发表的美国哈佛-MIT Broad研究所教授Eric Lander撰写的一篇关于CRISPR基因编辑技术发现史的综述文章,文章甫一发表就激起了文章中的一些当事人强烈的反应,其中CRISPR技术的开拓者和发明人之一Jennifer Doudna就公开对一些事实进行了驳斥。回顾CRISPR基因编辑技术发现史,这是一篇无法绕过的文章,今日重新发布,温故知新,对关键论文的截图进行了更新,以飨读者!
补充:据Nature网站报道,Doudna 在接到采访电话时的反应是“I grew up in a small town in Hawaii and I never in a 100 million years would have imagined this happening,” says Doudna. “I’m really stunned, I’m just completely in shock.” “I know so many wonderful scientists who will never receive this, for reasons that have nothing to do with the fact that they are wonderful scientists,” Doudna says. “I am really kind of humbled.”(https://www.nature.com/articles/d41586-020-02765-9)另据,UC Berkeley的诺奖新闻发布会的信息,Doudna 在回答记者问题环节提到她是从Nature的这位记者(Doudna的好朋友)这里得知她获奖的消息,此前她在深睡,手机静音中。
Doudna 在UCB举行的诺奖新闻发布会上截图
在Science的新闻报道中,哈佛大学的George Church教授表示诺奖委员会做出了一个非常棒的选择(“a really great choice”),不认为自己和张锋应该出现在获奖名单中。Church教授认为Charpentier和Doudna是做出了发现(“made a discovery”),这是诺奖委员会更看重的,而他和张可称之为发明家(“inventor”)。对于张,Church教授认为他还年轻,非常具有创造性,张锋将来还会有机会的。 (https://www.sciencemag.org/news/2020/10/crispr-revolutionary-genetic-scissors-honored-chemistry-nobel)
CRISPR英雄谱
原文丨埃里克·兰德(Eric Lander)
小鹿/译

Eric Lander教授第一时间发出的祝贺
摘要
三年之前,科学家们宣布,CRISPR技术能够对真核活细胞进行精准与有效的基因组编辑。自此,这项技术手段已然震撼了科学界,数以千计的实验室正在将其运用于从生物医药到农业的各个领域。然而,从一种奇特的细菌重复序列现象的发现开始,到确认这种现象为适应性免疫系统,进而对它的生物功能特性的了解,直至开发为一项基因工程的技术,这此前二十载相关的研究历程却不为人所知。本文正是着眼于填补这段科学历史的空白,它讲述的是观念的演化历史和先锋人物的传奇故事,并且从中获得关于支撑科学发现的优秀科研环境的启迪。
前言
很难想起曾经有哪一次科学革命像CRISPR这般如此迅速地改变生物学界。仅仅三年之前,科学家宣布,CRISPR系统,即细菌通过纪录和精准攻击入侵病毒的DNA序列而进行自身防御的适应性免疫系统,可以利用转化为一项简单而有效的技术在哺乳动物和其它生物体的活体细胞内进行基因组编辑。CRISPR即此被全球数以千计的实验室运用于广泛的领域,如创建人类遗传疾病和癌症的复杂动物模型;在人类细胞内进行全基因组筛选从而精确定位作用于生理过程的具体基因;开启或关闭某个特定基因的作用;改变植物的基因。CRISPR有可能用于改变人类生殖系统的前景引发了全球范围的争论。
虽然我还没见过没有听说过CRISPR的分子生物学家,但是,你如果问他们到底这项科学革命是如何发生的,他们往往一头雾水。免疫学家彼得・梅达沃爵士(Sir Peter Medawar)有云:“科学的历史会让大多数科学家感到无聊透顶”(Medawar,1968)。的确,科学家总是义无反顾地专注于未来。一旦一项事实在科学上得到确立,那么通向发现此事实的迂回路径则被归为无关紧要的轶闻。
然而,科学突破背后的科学家的故事可以使我们对于善于激发生物医药进步的这种神奇的科研环境有更多了解:有关灵感与规划,纯粹好奇心与实践运用, “无假设驱动(Hypothesis-free)”与“假设驱动(Hypothesis-driven)”的研究方法,个人与团队,新颖的视角和深厚的专业知识在其中发挥的各自功能。这些理解对于政府和基金会都尤为重要,因为仅在美国,这些组织就在生物医药研究领域一共投资了超过四百亿美元。对于常常把科学家想象为离群索居于实验室的孤独天才的普通公众,这些理解也同样重要。此外,对于正在接受科学训练的科学学徒而言,能对科学职业生涯有一个现实的图景式的把握,并将其作为导向和激励,更是十分有益。
在过去的几个月里,我一直试图理解CRISPR背后的20年的前程往事,这其中包括科学观念的历史和科学家的个人经历。本文的视角是建立在已发表的论文,个人访谈和其它材料(含期刊拒绝信)的基础之上的。最后,我尝试从其中得出一些普遍的经验。(作为背景,图1提供了一个对II型CRISPR系统的简要概述,正是这一种类被转化利用于基因组编辑。)
本文的关键内容是描述了一群踌躇满志的科学家偕同他们的合作者和其它未能详述的贡献者一起发现了CRISPR系统,揭开它分子机制的面纱,并将它转化利用为生物与生物医药研究的强大工具。他们共同列名于CRISPR英雄谱。
CRISPR的发现
故事开始于西班牙白色海岸上的地中海港口圣波拉(Santa Pola),那里的美丽的海岸与广阔的盐沼地几个世纪以来吸引着度假客、火烈鸟和盐业生产商(这个故事的地理如图2所示)。Francisco Mojica就在附近长大,常常光顾这片海滩,自然而然,当他于1989年开始在位于海岸上端的阿利坎特大学(University of Alicante)作博士研究的时候,他加入了一个研究地中海嗜盐菌(Haloferax mediterranei)的实验室,这是一种从圣波拉的沼泽分离出的具有极端耐盐性的古细菌。他的导师发现,培养基的盐含量似乎会影响限制性内切酶切割此微生物的基因,Mojica于是开始鉴定这一异种片段。在他检查的第一个DNA片段里,Mojica发现了一个奇怪的结构,即一个近乎完美的大致呈回文式对称、有30个碱基并被36个碱基的间隔隔开的多拷贝重复序列,而它与任何已知的微生物的重复序列家族都不相同(Mojica 等,1993)。
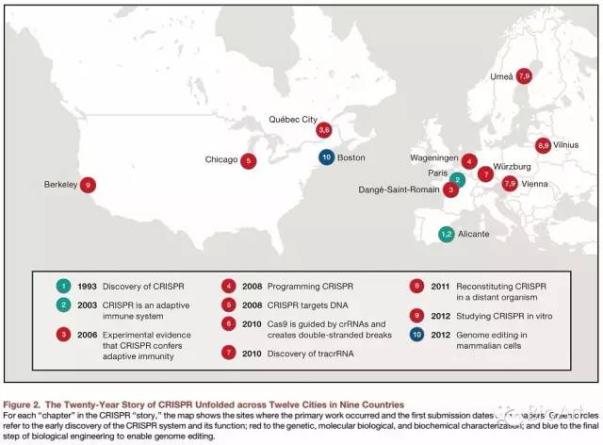
图2
这个28岁的研究生被此深深吸引,并且将他接下来的十年学术生涯贡献给了破解这一神秘现象。他不久在相近的沃氏嗜盐富饶菌(H.volcanii)和关系更远的嗜盐古菌中还发现类似的重复序列。在梳理科学文献过程中,他发现了这一现象与真细菌的关联:一个日本研究组(Ishino et al., 1987)的一篇论文提到在大肠杆菌(Escherichia coli)中的一个重复序列有相似的结构,虽然与嗜盐菌(Halofarax)的重复没有序列的相似性。论文的作者们对这一现象并未深究,但是Mojica意识到,在关系如此远的微生物上存在如此相似的结构一定意味着一个尚未被发现的原核细胞里的重要功能。在去牛津作短暂的博士后研究之前,他写了一篇论文报道了这一新的重复序列类别(Mojica 等, 1995)。
Mojica之后返回阿利坎特大学担任教职。由于学校缺乏启动科研基金和实验室空间,他只好转而通过生物信息学的方法来研究这种奇怪的重复,并将其命名为short regularly spaced repeats (SRSPs)。这一名称在他自己的建议下后来改为“聚集的规律插入间隔回文重复”(Clustered Regularly Interspaced Palindromic Repeats, CRISPR)(Jansen 等, 2002; Mojica 和 Garrett, 2012)。
到2000年,Mojica已在20个不同的微生物上发现了CRISPR基因位点,其中包括结核分枝杆菌(Mycobacterium tuberculosis),艰难梭菌(Clostridium difficile),和鼠疫杆菌(Yersinia pestis)(Mojica 等, 2000).。在两年的时间内,研究人员已经将这相关微生物的名录翻倍并且列录了基因位点的关键特征,包括存在近亲缘的特定的“CRISPR关联基因”(cas基因),这通常认为与他们的功能相关(Jansen 等, 2002)。(列表1列举了CRISPR系统的最新分类。)
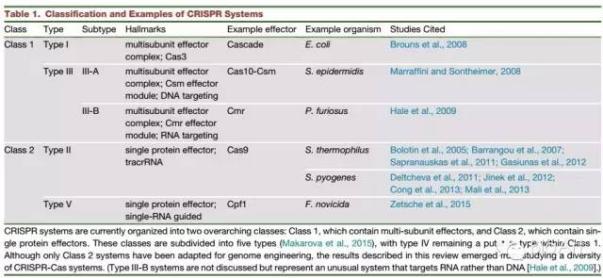
表1
但是,CRISPR系统的功能究竟是什么呢?各种假设层出不穷:比如设想与基因调控,复制分区,DNA修复还有其它功能有关。然而大多数的这类假设都没有证据支持,它们一个一个都被证伪了。如同CRISPR的发现一样,重要的睿见来自于生物信息学。
CRISPR是一种适应性免疫系统
2003年8月的假期,Mojica 避开圣波拉的酷暑,躲在阿利坎特的空调办公室里。如今已然作为初兴的CRISPR领域的领军人物,他将目光从重复序列转向分隔它们的间隔序列。使用文字处理器,Mojica 不辞辛劳地抽出每一个间隔并将其插入BLAST软件来搜索与其它任何已知DNA序列的相似性。虽然他尝试这种方法失败过,但是DNA序列数据库在不断扩大,这一次他成功掘到金矿了。在他最近从一种大肠杆菌菌株测序到的一个CRISPR基因位点上,其中一个间隔与一种P1噬菌体的序列相匹配,而这一噬菌体可以感染多种大肠杆菌菌株。然而,携带这一间隔的菌株已知对P1感染具有抵抗力。那一周结束时,他已经检索了4500个间隔。其中88个间隔与已知序列相似,三分之二同携带间隔的微生物相关的病毒或接合质粒相匹配。Mojica意识到CRISPR基因位点储存了用于为保护微生物抵抗感染的适应性免疫系统所需的信息。
Mojica 于是和同事们一起外出饮干邑白兰地庆祝,并在第二天清晨开始撰写相关论文。如此竟开始了长达18个月的痛苦煎熬。认识到这一发现的重要性,Mojica 将论文投给了《自然》(Nature)。2003年11月期刊在未征询外部评审的情况下拒绝的论文的发表。难以理解的是,编辑声称论文的关键论点属于已知范畴。2004年1月,《美国国家科学院院刊》(PNAS)的决定是这篇论文缺乏“充足的新颖观点和重要性”因而不够资格送审。《分子微生物学》和《核酸研究》(NAR)也相继地拒绝发表。此时,绝望而又担心被别人抢先一步发表的Mojica将论文投给了《分子演化杂志》。在经过12个多月的审稿和修订,这篇宣布CRISPR可能功能的论文终于在2005年2月1日发表了(Mojica et 等, 2005)。

与此同时,CRISPR正是另一个意想不到的地点的研究人员的关注焦点,那是巴黎向南30公里以外,法国国防部的一个研究部门。Giles Vergnaud 是在巴斯德研究所受训的人类遗传学家,他的博士和博士后研究都受到法国武器装备总局的资助。他在1987年完成博士后研究之后加入法国国防部并建立它的第一个分子生物学实验室。在以后的十年间,Vergnaud继续着人类遗传学的研究工作。但是,情报部门在九十年代后期关于伊拉克的萨达姆·萨达姆政权正在发展生物武器的报告引起关切之后,国防部在1997年要求Vergaud和他的团队将研究重心转向法医微生物学,籍此发展基于微生物种类间细微遗传学差别来追踪病原体来源的手段。在与附近的巴黎第十一大学的遗传与微生物学研究所的联合实验室,他开始使用串联重复序列多态性,这一法医学人类DNA指纹图谱的主要工具,来绘制导致炭疽病(anthrax)和鼠疫的细菌种类。
法国国防部拥有一批特殊的来自1964至1965年越南鼠疫爆发期间的61件鼠疫杆菌样本。Vergnaud发现这些密切相关联的分离株在串联重复基因位点上是一致的,除去一个位置是例外,即由他的同事Christine Pourcel所发现的CRISPR基因位点。它们的品系由偶尔出现新间隔所区分,而这些间隔无不是在CRISPR基因位点的前段末端(Pourcel 等,2005)。值得注意的是,其中许多的新间隔与存在于鼠疫杆菌基因上的原噬菌体(prophage)相配。作者们推断,CRISPR基因位点是在执行防御机制,用诗化的语言来说,就是“CRISPR可能重现‘过往遗传攻击’的记忆。”Vergnaud试图发表他们发现的努力和Mojica遇到了相同的阻碍。论文被《美国国家科学院院刊》、《细菌学学刊》、《核酸研究》以及《基因组研究》相继退稿,直到在2005年3月1日的《微生物学》上发表。

最后,第三个研究者Alexander Bolotin,供职于法国国家农业研究院的俄裔微生物学家也在2005年9月的《微生物学》上发表了关于CRISPR 起源于染色体之外的论文(Bolotin等,2005)。由于他的论文之前被另一家期刊拒绝,所以事实上是在Mojica的2005年2月论文发表之后一个月后才得以提交。值得注意的是,Bolotin第一个提出了CRISPR是如何提供免疫功能的设想,他推测来自CRISPR基因位点上的转录物是凭借反义RNA对噬菌体基因表达的抑制来工作的。这一假设虽然听上去合理,但是很快会被证伪。

CRISPR提供适应性免疫功能并且使用核酸酶的实验证据
如同Mojica,Philippe Horvath可能找不到一个更有地方特色或者说更无趣的论文题目了。身为斯特拉斯堡大学(University of Strasbourg)的博士生,他的研究对象是一种用于德式腌白菜(sauerkraut)生产的乳酸菌, 这种腌白菜正是阿尔萨斯(Alsatian)风味菜品腌白菜猪肉土豆(choucroutegarnie)的主要配料。出于他对食品科学的兴趣,Horvath跳过博士后研究而在2000年加入了罗地亚食品公司(Rhodia Food),一家位于法国西部当热圣罗曼(Dange-Saint-Romain)的细菌发酵剂生产商,在那里建立这家企业的第一个分子生物学实验室。这家公司之后由丹麦公司丹尼斯科(Danisco)收购,而丹尼斯科自己则在2011年被杜邦公司(DuPont)收购了。
罗地亚食品公司对于Horvath的微生物学专长十分感兴趣,因为其它的乳酸菌诸如嗜热链球菌(Streptococcus thermophiles)一直用于乳制品的生产,比如酸奶和芝士。Horvath的目标包括开发出基于DNA的技术手段,以精确识别菌株并且战胜频繁性噬菌体感染这一困扰着乳制品发酵所用的工业发酵剂生产的痼疾。因此,理解特定嗜热链球菌菌株是如何抵御噬菌体攻击而自我保护的机制则具有了科学和商业上的双重价值。
在2002年下半年的一次乳酸菌主题的荷兰会议上得知CRISPR之后,Horvath就开始使用这一手段来鉴定他的菌株的基因型。到了2004年下半年,他注意到间隔与抵抗噬菌体能力之间存在关联,相同的发现在几个月之后正是由Mojica和Vergnaud发表的。2005年, Horvath和他的同事们,包括任职丹尼斯科美国分公司的新科博士Rodolphe Barrangou和魁北克城( Quebec City)的拉瓦尔大学(Universite Laval)的著名噬菌体生物学家Sylvain Moineau一同着手检验CRISPR是一种适应性免疫系统这一假设。巧合的是,Moineau也曾是在工业领域任职的科学家。他在拉瓦尔取得食品科学博士学位,研究乳酸菌,并且在返回拉瓦尔任教职之前在联合利华公司(Unilever Corporation)工作。他自2000年以来曾同罗地亚食品公司保持合作。
利用一种特征显明而对噬菌体敏感的嗜热链球菌菌株和两种噬菌体,研究人员通过遗传筛选的方法来分离出对噬菌体有抵抗力的菌株。并非包含传统的抵抗性突变(诸如在噬菌体侵入所需要的细胞表面受体上的突变),这种具有抵抗力的菌株在它们的CRISPR基因位点上获得了源于噬菌体的序列(Barrangou 等,2007)。此外,多重间隔的插入与增强的抵抗力相关。它们正是在这一过程中获得了免疫功能。
他们还研究了其中的两个cas基因的作用:即cas7和cas9。细菌需要cas7来获得抵抗力,但是这些携带噬菌体源的间隔却不需要这个基因来保持抵抗力,这就说明cas7协助产生新的间隔和重复,但它并不涉及免疫功能本身。与此相反,cas9则对抵抗噬菌体的能力产生必不可少,因它的序列包含两种核酸酶模块(HNH和RuvC)而它的产物继而被认为对核酸进行了切割(Bolotin等,2005; Makarova等,2006);并且,cas9蛋白是组成细菌免疫系统的活跃成分。(提示:在早期的CRISPR文献中,如今大名鼎鼎的cas9基因被称作cas5或者csn1。)
最后,他们发现,能够克服基于CRISPR免疫功能的稀有噬菌体分离株在它们的基因里携带有单一碱基变化,如此就改变了本来与间隔相配的序列。这样,免疫功能就是依赖间隔与目标物之间的精准DNA序列匹配来实现的。
设计CRISPR
John van der Oost于1989年在阿姆斯特丹自由大学(Free University of Amsterdam)获得博士学位,他最初志在解决世界清洁能源的需求而研究如何利用蓝藻来生产生物燃料。在返回阿姆斯特丹之前,他相继在赫尔辛基和海德堡研究细菌的代谢途径。1995年,瓦赫宁根大学(Wageningen University)向他发出终身教职的聘任邀请,但是条件是需要由他来壮大一个专攻极端条件生存的微生物的团队。Van der Oost 在德国期间已经听说过能够在黄石国家公园的温泉中繁衍的嗜热链球菌,于是对探索这些奇特微生物在代谢途径上的演化区别跃跃欲试。他开始和隶属于美国国家卫生研究所(National Institutes of Health)的国家生物科技信息中心(NCBI)的微生物进化和计算机生物学领域的专家Eugene Koonin合作。Koonin早已开始对CRISPR系统进行数据分类和分析,他在2005年的一次访问中将van der Oost领入当时还鲜为人知的CRISPR领域(Makarova等,2006)。
Van der Oost当时已从荷兰国家科学基金获得了主要资助。他决定在研究申请书的课题之外,将部分经费用作CRISPR的研究。(在五年之后的报告中,他强调了上述机构给予研究人员改变研究计划方向的自由这一政策的有益价值。)
他和同事们将一种大肠杆菌的CRISPR系统嵌入另一缺乏这一自身内源性系统的大肠杆菌品系。这样就可以使他们可以在生物化学上鉴定一组拥有5个cas蛋白的复合体,它被称作Cascade(Brouns等,2008)。(大肠杆菌拥有更复杂的1类,I 型CRISPR系统,其中cas9的功能是由Cascade复合体联同核酸酶cas3一起来实现的。见列表1。)
通过将每一个组成部分逐一剔除,他们证明Cascade是将经由CRISPR基因位点转录的一个长前体RNA接入61个核苷酸长的CRISPR RNA(crRNAs)所必需的。经过对一组与Cascade复合体一同纯化的crRNAs进行克隆和基因测序,发现它们都从8个碱基的重复序列开始,紧随其后的是完全间隔和新重复区的出现。这一发现支持了先前的假设,即重复序列的回文(palindromic)结构特性导致crRNA中次级结构的形成(Sorek等,2008)。
为了证明crRNA序列是产生基于CRISPR的抵抗力的原因,他们着手创造首个人工CRISPR排列,设定使CRISPR将λ噬菌体的四种基本基因作为定向靶位。正如他们所料,携带新的CRISPR序列的品系对噬菌体呈抵抗性。这是有史以来第一个直接由程序设计的基于CRISPR的免疫,对细菌而言犹如流感疫苗。
这些实验结果暗示,CRISPR的目标不是RNA(Bolotin所设想的),而是DNA。研究人员设计了两种版本的CRISPR排列,一种是反义链方向的(与mRNA和DNA位点的编码链互补),另一种是正义链方向的(仅仅与另一条DNA链互补)。虽然,间隔在有效性上各有差别,但是实验在正义链方向的版本上起了效果这一事实有力说明目标不是mRNA。但是,它不是直接证据。在《科学》的编辑们针对论文作出坚定论断保持谨慎的要求下,van der Oost将CRISPR定位DNA为目标物的观点以“猜想”的方式提出。

CRISPR的靶标是DNA
Luciano Marraffini正在芝加哥大学完成博士学位的研究,研究方向是葡萄球菌(Staphylococcus),此时从系里的噬菌体遗传学世界权威Malcolm Casadaban那里了解到CRISPR。Casadaban于2005年旋即看出CRISPR有可能是适应性免疫系统这一发现的重要程度,而且对所有对此表现出兴趣的人都谈论CRISPR。和许多噬菌体研究领域的科学家一样,Marraffini坚信CRISPR不是由RNA干扰来作用的,因为这一机制对于克服发生在噬菌体感染过程中的爆发式增长是无能为力的。他推断,CRISPR必然对DNA进行了切割,这个功能就好像限制性内切酶的作用。
Marraffini本来热切希望加入世界上为数不多的几个正在研究CRISPR的研究团队来做博士后的研究,可是由于他的妻子在库克县的伊利诺伊(Cook County, Illinois)刑事法庭有一个做翻译员的好工作,他感觉自己必须留在芝加哥。他说服西北大学(Northwestern University)的生物化学家Erik Sontheimer让自己加入他的实验室研究CRISPR,Sontheimer一直从事RNA剪接和RNA干扰的工作。
早在搬去西北大学之前,Marraffini在完成博士研究同时就开始了关于CRISPR的工作,研究葡萄球菌(Staphylococcus)的CRISPR系统是否可以阻止质粒接合。他注意到,一种表皮葡萄球菌(Staphylococcus epidermidis)拥有这样一个间隔,它与来自具有抗生素耐药性的金黄色葡萄球菌的质粒上存储的切口酶(nickase,nes)基因的某区域相匹配。他证明了,这些质粒不能转化表皮葡萄球菌,而中断质粒内nes序列或者它在CRISPR基因位点上的相配间隔的两者之一都会取消干扰功能(Marraffini 和 Sontheimer,2008)。显而易见,CRISPR如同阻止病毒一样阻止了质粒。
Marraffini和Sontheimer一度考虑在试管里重组CRISPR系统来证明它可以切割DNA。但是表皮葡萄球菌的系统太过复杂,它有9个cas基因,并且它的基因特点还完全未被掌握。于是,他们将目光转向分子生物学。他们聪明地对CRISPR系统定向射靶的质粒中的nes基因进行了改变,就是通过在它的序列中间嵌入一个自我剪接的内含子。如果CRISPR是以mRNA为目标的话,那么这一变化就不会影响干扰功能,因为内含子序列会被剪除。而如果CRISPR是以DNA为目标的话,那么这种嵌入就会取消干扰功能因为间隔将不再匹配。结果已经清楚:CRISPR的目标是DNA。
Marraffini和Sontheimer认识到CRISPR在本质上是一种可编辑设定的限制性内切酶。他们的论文则第一个清楚提出了预测:CRISPR可以被转而用于在异源性系统内进行基因组编辑。“从实践的观点来看,” 他们宣布,“可以对包含任何已知24至48个核苷酸目标物的DNA进行可定位灭除的导向能力具有可观的功能性用途,特别是如果这个系统还可以在原来的细菌或古菌环境以外发挥作用。”他们甚至提交了涉及使用CRISPR在真核细胞内切割和纠正基因位点的专利申请,然而由于尚缺乏实验证明而最终放弃了申请(Sontheimer和Marraffini,2008)。
Cas9是由crRNAs指引并且在DNA中产生双链裂断
在2007年影响深远的研究确认了CRISPR是一种适应性免疫系统之后(Barrangou等,2007),Sylvain Moineau继续与丹尼斯科公司合作来弄清CRISPR切割DNA的机制。
问题是CRISPR在通常情况下总是如此高效,使得Moineau和同事们没法轻易观察到入侵的DNA是如何被消灭的。但在研究嗜热链球菌的质粒干扰过程中他们受到了幸运女神的眷顾。研究人员发现了少量品系,它们的CRISPR对依靠电穿术(electroporation)转化的质粒仅仅可以提供部分防卫。在其中一个低效的品系的细胞内,可以看见呈线性的质粒依旧存在。一定程度上,质粒干扰的过程能够被减缓到使CRISPR活动的产物可以被观察到(Garneau等,2010)。
这一品系使他们得以研究切割的过程。与他们先前的结果一致(Barrangou等,2007),这次的结果显示质粒的切割依赖cas9核酸酶。当对线性化的质粒进行测序时,他们在PAM(proto-spacer adjacent motif)的上游发现了单个精准平端切割的三核苷酸,PAM是一种关键性序列特征,而先前的论文对它的作用已有描述(Deveau等,2008;Hovath等,2008)。将分析的范围扩展之后,结果表明病毒的DNA也是在与PAM序列有关的相同位置被精准切割的。此外,匹配同一目标的不同间隔的数量与切口数量一致。
实验结果确定无疑地表明Cas9的核酸酶活性是在精准的点位上对DNA进行切割的,而这些点位则由crRNA的特定序列编码。
tracrRNA的发现
即使在对CRISPR-Cas9系统的密集研究之下,组成这个难题一块拼图却仍然缺失,就是后来被称作反式作用CRISPR RNA(tracrRNA)的一个小RNA。事实上,它的发现者Emmanuelle Charpentier(诺奖得主之一)和Jörg Vogel并不是专门研究CRISPR系统的;他们仅仅试图鉴定微生物RNA。
Charpentier于1995年从巴斯德研究所(Pasteur Institute)获得博士学位,之后在纽约做了6年的博士后研究,然后相继于2002年在维也纳大学和2008年在瑞典的于默奥大学(UmeåUniversity)建立自己的实验室。她在化脓性链球菌(Streptococcus pyogenes)里发现了一种不寻常的可控制其传染力的RNA之后(Mangold等,2004)开始对鉴定更多微生物内的调控性RNA发生了兴趣。她用生物信息学的程序检索化脓性链球菌的基因区间,设想它们可能编码非编码RNA。她发现了若干候选区间,包括在CRISPR基因位点附近的一个,——但是缺乏关于这些RNA的直接信息,就太难继续研究了。
当Charpentier在威斯康辛的麦迪逊(Madison, Wisconsin)召开的2007年RNA学会上遇见Vogel时,解决方法出现了。在德国受训为微生物学家的Vogel在乌普萨拉(Uppsala)和耶路撒冷(Jerusalem)做博士后研究期间开始专攻在病原体内寻找RNA,这一工作持续至2004年他在位于柏林的马普感染生物学研究所建立自己的研究小组时。(5年之后,他转去乌兹堡(Würzburg)领导一个传染病研究中心。)随着“下一代测序”技术的到来,Vogel意识到大规模平行测序将使得绘制任何微生物转录组的整体图录成为可能。他当时已经将这种方法用于导致胃溃疡的细菌——幽门螺杆菌(Sharma等,2010),同时正在用于其它细菌。Charpentier和Vogel决定将目标瞄准化脓性链球菌。
此方法产生了一个惊人的结果:仅次于核糖体RNA和转运RNA之后数量第三多的RNA转录本属于一类新的小RNA,它是从紧挨着CRISPR基因位点(就是那个曾引起Chapentier注意的区间)的序列转录过来的,并且它有25个与CRISPR重复序列近乎完美地互补的碱基。这一互补性说明tracrRNA和crRNA的前体一起杂交并且被RNaseIII切割处理成为成熟产物。遗传删除实验也证实了这个观点,即tracerRNA对处理crRNA是必不可少的,因而就对CRISPR作用也是必不可少(Deltcheva 等, 2011)。

之后的研究揭示了tracerRNA还有另一个关键作用。接下来的生物化学研究表明tracerRNA不仅仅涉及处理crRNA,它还对Cas9核酸酶复合体剪切DNA是必需的(Jinek 等, 2012; Siksnys 等, 2012)。
在远缘物种内重建CRISPR
Virginijus Siksnys成长于苏联时代的立陶宛(Lithuania),从维尔纽斯大学(Vilnius University)毕业之后于1980年代早期前往莫斯科国立大学(Moscow State University)攻读博士学位,研究酶动力学。这之后他回到家乡维尔纽斯加入应用酶学研究所,从事当时热门的限制性内切酶领域的研究。20年之后,他对于研究限制性内切酶已经感到厌烦。Horath,Barrangou和Moineau的2007年论文重燃了他对于细菌抵御外部DNA的屏障的兴趣。作为化学家,他意识到想要理解CRISPR,就必须在体外重建它。
他的第一步就是要测试他是否已经获取了所有的必要组成部分。他和合作者们着手观察来自嗜热链球菌(S. thermophiles)的CRISPR基因位点是否能够在一个远缘的微生物大肠杆菌(E. coli)内被重建成为完全发挥作用的形态。令他们感到欣喜的是,他们发现转入整个CRISPR基因位点就足以实现对质粒和噬菌体DNA的靶向干扰(Sapranauskas 等,2011)。他们用异源系统还证明了Cas9是干扰活动唯一必需的蛋白,而它的Ruvc和HNH核酸酶结构域都是必不可少的。
随着CRISPR-Cas9干扰系统的必要与充分的组成部分——Cas9 核酸酶,crRNA和tracrRNA都被发现了,这一领域的研究到达了一个关键性的里程碑。这个系统依靠尖端的生物信息学、遗传学和分子生物学已经被化整为零地完全掌握了。如今是时候将方向转向精确的生物化学实验来证实并且在试管里拓展这些结论了。
在试管里研究CRISPR
利用它们在大肠杆菌里的异源表达系统,Siksnys和同事们通过使用链酶亲和素标记物标记Cas9来纯化嗜热链球菌的Cas9-crRNA复合体,并在在试管内观察它的活动(Gasiunas 等,2012)。结果表明,复合体能够在体外切割DNA目标,创造出一个距离PAM序列恰好3个核苷酸的双链断裂——正好与Moineau及其同事在细菌体内观察到的现象一致。最重要的是,实验证明他们能够在CRISPR阵列里对Cas9和专门设计的间隔序列一同改编,从而实现在试管内对选择目标位置的切割。通过转变HNH与RuvC核酸酶结构域的催化残基,他们还证明了前者切割与crRNA互补的基因链,而后者则切割与之相对的那条链。他们同时又证明了crRNA可以被修剪至只剩20个核苷酸而仍然保持有效切割力。最后,Siksnys证明这个系统还可以用第二种方法重建——就是把纯化的His标记的Cas9、在试管内转录的tracrRNA和crRNA,还有RNase III混合起来——而上述两种RNA对于Cas9切割DNA都是必不可少。(最终他们在修订后的论文里删除了第二种重建方法,但是在他们已发表的提交于2012年3月的美国专利申请书中报告了所有的研究内容[Siksnys 等,2012])。

与此同时,Charpentier已经开始与维也纳的一个同事一起对CRISPR进行生物化学鉴定。在2011年3月在波多黎各(Puerto Rico)举行的美国微生物学学会会议上报告她关于tracrRNA的研究的时候,她遇见了Jennifer Doudna(诺奖得主之一),加利福尼亚大学的世界著名结构生物学家和RNA专家。夏威夷长大的Doudna在哈佛大学获得博士学位,与Jack Szostak合作将一个RNA自我剪接内含子重塑为具有复制RNA模版能力的核酶。她在之后跟科罗拉多大学的Tom Cech做博士后研究时解决了核酶的晶体结构问题。在她自己的实验室(起先于1994年在耶鲁建立,之后于2002年在伯克利),她鉴定了多种现象下的RNA蛋白复合体,诸如与内部核糖体入口位置有关和microRNA的加工有关等。她一直使用晶体学和冷冻电镜来解决I型CRISPR系统的Cascade复合体的组成部分的结构问题,这是一种用于诸如大肠杆菌的较为复杂的系统。
两位科学家于是决定联手。她们使用重组Cas9(来自在大肠杆菌里表达的化脓性链球菌[S. pyogenes]的基因)和体外转录的crRNA及tracrRNA(Jinek 等,2012)。和Siksnys一样,她们也证明了Cas9可以在体外切割纯化的DNA,它可以和专门设计的crRNA一起被设计,两种核酸酶结构域分别切割相对的两条DNA链,并且crRNA和tracrRNA对Cas9发挥作用都是必需的。此外,她们证明两种RNA在被融合为单一的导向RNA(sgRNA)时也可以在体外发挥作用。在经过其他科学家的修改而变得可以更高效地在体外作用之后,sgRNA的概念在基因组编辑领域被广泛使用。

Siksnys于2012年4月6日向《细胞》提交了论文。6天之后,在没到外部评审的情况下就被期刊拒绝了。(事后,《细胞》的编辑承认这篇论文其实是非常重要的。)Silsnys于是做了此论文的精炼版本,于5月21日将其投稿给了《美国国家科学学院院刊》,得以于9月4日在线发表。Charpentier和Doudna的合作论文的运气则要好得多。在Siksnys的论文提交之后的2个月,她们的论文于6月8日提交给了《科学》,顺利通过评审并于6月28日在线发表。

两组研究团队都清楚地认识到CRISPR对于生物技术的潜在价值。Siksnys宣称:“这些发现为构造普遍可设计的RNA引导的DNA核酸内切酶铺平了道路。”而Charpentier和Doudna则提到:“利用这一系统来进行可设计的基因组编辑的潜力。”(几年之后,Doudna让世界的注意力里投向编辑人类生殖系统这一前景所引起的重要社会性问题。)
在哺乳动物细胞内进行基因组编辑
在1980年代后期科学家们设计出一种可以在活细胞内改变哺乳动物基因的方法,这彻底改变了生物医学研究,包括使得在老鼠的胚胎干细胞内的特定位置嵌入DNA并且培育出携带这一遗传改变的后代成为可能(综述见Capeccchi,2005)。虽然这个方法是革命性的,但是过程却是低效的,因为它需要通过筛选识别出那些百万分之一的细胞,就是在其内通过同源重组与由实验者提供的修改过的版本之间交换了一个基因。1990年代中期,哺乳动物学家在酵母遗传学的观察基础上发现在某个基因位点上引进一个双链断裂可以极大地增强同源重组和由非同源末端接合导致的小缺失的发生频率(综述见Haber,2000和Jasin与Rothstein,2013)。他们意识到,高效基因组编辑的秘诀在于找到一种可以在任何想要的位置制造出双链断裂的可靠手段。最初的普遍策略是使用锌手指核酸酶(ZFNs)——一种由一个锌手指DNA结合域和一个取自限制性内切酶的DNA切割结构域所组成的融合蛋白,它可以结合并切割基因组位点(Bibikova 等,2001)。不久科学家们就在果蝇和小鼠身上证明了ZFNs依靠同源重组对于具体位置上的基因组编辑的用途(Bibikova 等,2003;Porteus和Baltimore,2003)。到2005年,桑加莫生物科学公司(Sangamo Biosciensces)的研究小组报告了针对在人类细胞系上造成重度联合免疫综合症的基因的突变进行了成功的修正(Urnov 等,2005)。然而,塑造能够可靠辨认具体位置的ZFNs的过程是缓慢而费力的。更好的方法则出现在2009年下半年,两个研究小组描述了一组来自于植物病原体黄单胞菌(Xanthomonas)(Boch 等,2009;Moscou和Bogdanove,2009),叫做TALEs的特殊转录激活蛋白,TALE使用一个特定的模块密码来瞄准具体的DNA序列。但是,这一方法还是需要可观的工作量,因为针对每个目标都要求设计一个新的蛋白。
自从Marraffini和Sontheimer的2008年论文证明了CRISPR是一种可设计的限制性内切酶以来,研究人员已经意识到如果它可以在哺乳动物细胞内发挥作用的话,CRISPR也许就能为具体基因位点的切割和编辑提供强大的工具。但是,这个“如果”是非常关键的。相对于微生物,哺乳动物细胞拥有不同的内部环境,它们的基因要大1000倍,位置在细胞核内,并且嵌在一个精致复杂的染色质结构内。转入诸如自我剪接II类内含子的其它简单微生物系统已经失败了,而使用核酸来瞄准基因位点的尝试也问题重重。CRISPR究竟能否被设计构造成为一种用于编辑人类基因组的强力工具?直到2012年9月,专家们是持怀疑态度的(Barrangou,2012;Carroll, 2012)。
张锋11岁时从中国石家庄搬到爱荷华州的得梅因(Des Moines, Iowa)。他在一次周六兴趣课程中被分子生物学深深吸引,在16岁时就在一家当地的基因疗法实验室每周工作20小时。在哈佛念本科的时候,他因为室友患了重度抑郁症的事而开始对大脑发生兴趣,后来他到斯坦福跟随神经生物学家兼精神病学家Karl Deisseroth攻读博士学位,期间他们(联同Edward Boyden)发展出了光遗传学(optogentics),这是一项革命性的技术,依靠此技术可以通过光束来触发携带微生物光敏感通道蛋白的神经元。在波士顿身为独立研究员期间(先后在哈佛和麻省理工学院的脑及认知科学系还有博德研究所[Broad Institute]作初级研究员),张致力于进一步扩展研究神经生物学的分子工具。在开发出一种使用光来激活基因表达(通过把一个DNA结合结构域和一个转录激活结构域同两个在光条件下相互结合的植物蛋白偶联)的方法之后,他开始探索一种用来设计转录因子的普遍方法。在TALEs被解码之后,张与他的合作者Paola Arlotta和George Church(此外还有一个来自桑加莫生物科学公司的研究小组)都成功地将TALEs改造用于哺乳动物,使得精准激活、抑制和编辑基因成为可能(张等,2011;Miller等,2011)。然而,张锋并没有停止寻找更好的方法。
2011年2月张听了哈佛的微生物学家Michael Gilmore关于CRISPR的报告就立刻被它吸引住了。他次日飞赴迈阿密参加一个学术会议,却闷在酒店房间里一头扎进CRISPR的文献资料。回来之后,他就着手创造嗜热链球菌的Cas9版本用于人类细胞(用优化密码子和一个核定位信号)。到2011年4月,他已经发现,通过表达Cas9基因和一个设计过的靶向携带荧光素酶的质粒的CRISPR RNA,他可以在人胚胎肾细胞(HEK)内降低荧光度。但是,效果还是不尽如意。
接下来的一年间张一直在优化这个系统。他探索着可以增加进入细胞核的Cas9比例的方法。当他发现嗜热链球菌Cas9在细胞核内并非平均分布(它在核仁处聚集),他就测试其他的选择,发现化脓性链球菌(S. pyogenes)的Cas9分布地更均匀。他发现哺乳动物细胞虽然缺乏细菌的RNaseIII,但是仍然可以处理crRNA,尽管是用和在细菌里不同的方式。他测试了tracrRNA的各种同工型(isoform)来造鉴定在人类细胞内保持稳定的那一个。
到了2012年的中期,他已经获得了一个由三部分组成的强有效的系统,包含来自化脓性链球菌和嗜热链球菌两者之一的Cas9、tracerRNA和CRISPR阵列。以人和小鼠基因的16个位置为靶向,他证明了,高效而准确地改变基因是可能的——通过非同源重组的末端接合制造缺失和通过与修复模版进行同源重组来实现插入新序列。此外,多重基因可以通过设计CRISPR阵列,用彼此匹配的间隔来同时编辑。当Charpentier和Doudna的论文在那年初夏发表之时,他还用在她们的实验里描述的短sgRNA融合来测试一个两部分组成的系统。在体内环境下这一融合的效果很差,仅仅低效地切割了位点的一小部分,但是,他发现一个复原了3‘端发夹结构的全长融合可以解决问题(Cong 等,2013;Zhang,2012)。(张之后很快继续证明了CRISPR比原想的更加功能多样:它可以被用于在数周内创造遗传疾病和体细胞癌的复杂小鼠模型,也可以用于实施全基因组筛选来寻找某一生理过程中的必须基因——它的精确度还可以通过降低脱靶切割来提高。他和曾与van der Oost共事的计算机生物学家Koonin还将发现新的第2类CRISPR系统,包括那个与Cas9切割方式不同而且仅需要crRNA却不需要tracrRNA的核酸酶系统[Zetsche 等,2015]。)张于2012年10月5日提交了报告哺乳动物基因组编辑的论文,发表在2013年1月3日的《科学》上(Cong 等,2013);这篇论文将成为该领域内被引用次数最多的论文,而他的试剂由一家非营利性组织Addgene分发,在之后的3年间接受到25000次申请。

大约一个月后的10月26日,一个在基因组学和合成生物学领域极有专长并且曾与张有合作的才华横溢而非同寻常的哈佛大学资深教授George Church递交了他关于在人体细胞内进行基因组编辑的论文。自从1970年晚期跟随DNA测序先锋Walter Gibert在哈佛读研究生期间,Church就专注于发展大规模“阅读”和“书写”基因的强大技术——此外他还因具有挑衅性的建议,譬如使用合成生物学来复活长毛的猛犸象(wooly mammoths)和尼安德特人(Neanderthals)而引起社会争论。清楚张的研究,同时受到Charpentier和Doudna的启发,Church着手在哺乳动物细胞内测试crRNA-tracer融合。和张一样,他也发现短融合在体内环境是无效的,而全长融合则效果好。他瞄准7个位置,证明了非同源重组末端接合和同源重组的作用。他的论文和张的论文是相继发表的(Mali 等,2013)。(Church和其他研究者都在不久之后使用CRISPR来提升“基因驱动力”(gene drive)——合成基因在自然群体中可以迅速扩散,从而引发将其应用于控制携带疟疾的蚊子的惊喜和对于破坏生态系统的担忧。他还将寻求使用CRISPR在猪的基因组里灭活逆转录病毒来推动猪的器官移植给人。)
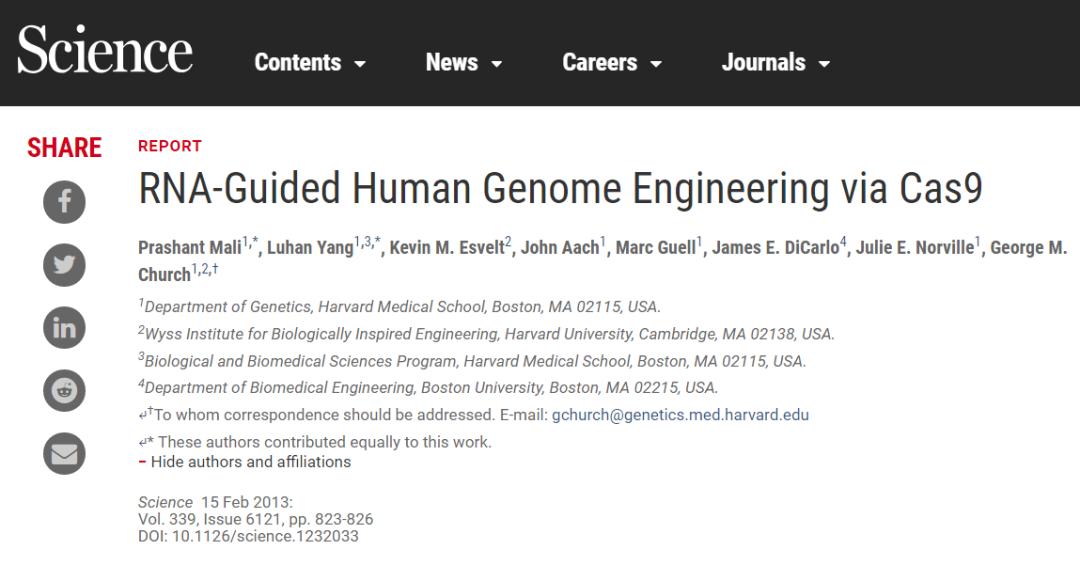
到了2012年的夏末,随着体外研究获得瞩目,而成功的体内基因组编辑的消息在发表之前就传播开来,好几个团队开始竞相开展基因组切割的概念验证实验,尽管不是基因组编辑。Doudna在Church的协助下提交了一篇论文,证明在一个基因组位点进行低效率的切割(Pandika 2014;Jinek 等,2013)。曾从事ZFNs和TALEs基因组编辑的韩国首尔国立大学(Seoul National University)的Jin-Soo Kim 教授报道了在两个位点进行切割(Cho 等,2013)。在以上两个例子中,切割都是低效的,因为sgRNA缺乏tracerRNA的关键性3‘端发夹结构。作为使用ZFNs和TALEs进行基因组编辑的领军人物,哈佛大学教授Keith Joung 则更进了一步。使用由他的合作者Church提供的全长sgRNA结构,Joung通过在斑马鱼的实验证实CRISPR可以在生殖系上有效地制造缺失(Hwang 等,2013)。这些短论文提交于2012年末,在张和Church的论文2013年1月初发表不久之后被接收,于1月末在线发表。
CRISPR迅速蹿红
2013年初,用谷歌搜索“CRISPR”开始井喷,这一趋势至今未减弱。在一年之内,研究人员已经报告了在多种生物内使用基于CRISPR的基因组编辑,包括酵母、线虫、果蝇、斑马鱼、小鼠和猴子。科学和商业上对其在人类医疗和农业生产的潜在应用的兴趣也开始上升,同时这个技术可能被用来培育设计婴儿的前景也引发了社会关注。
CRISPR的早期开拓者并未停止拓展边界,但是他们不再孤单。全世界的科学家都涌入了这一领域,他们是一批新的英雄队伍,他们进一步地阐明CRISPR的生物学,推进和扩展了基因编组辑技术,并且将它运用于广泛范围的生物学问题。在这篇文章的范围内公正描述他们的贡献是不能的;读者可以参考最近的综述(Barrangou和Marraffini,2014;Hsu 等,2014;van der Oost 等,2014; Sander和Joung,2014;Jiang和Marraffini,2015;Sternberg和Doudna,2015;Wright 等,2016)。
发现20年前的一处西班牙盐沼而曾经不为人知的微生物学系统如今却成了科学期刊特刊、《纽约时报》的头条、生物科技创业公司和国际伦理学峰会的焦点。CRISPR的时代已经到来了。
CRISPR的启迪
CRISPR的故事充满了关于产生科学进步的人文环境的启迪,与学术资助机构、一般公众和踌躇满志的研究人员相关。
其中最重要的一点是医学突破往往来源意想不到的地方。CRISPR的早期英雄们都不是专门研究编辑人类基因的,甚至都是不是研究人类疾病的。他们的动机混合了个人兴趣(弄清楚耐盐细菌内奇怪的重复序列)、军事上的特殊需要(防范生物战争)和工业应用(提高酸奶产量)。
这段历史还说明了基于大数据的“无假设驱动”式发现在生物学研究中愈发重要的位置。CRISPR基因位点,它的生物学功能和tracrRNA的发现都不是来自实验台上的实验,而是来自对大范围、常常是公开的基因数据库的生物信息学的开放式探索。“假设驱动”科学当然依旧是基本的研究方法,但是21世纪将会看到更多的是两种科学研究方法的结合。
如此众多的CRISPR英雄们都在科学生涯的开头(包括Mojica,Marraffini,Charpentier, Vogel还有张)于30岁之前就做出了他们各自的重要成果是很有启发意义的。年轻常常意味着愿意在未知的方向和看似无人问津的问题上冒险——和想要获得成功的动力。对于如今这个首次获得NIH资助的年龄已经升至42岁的时代而言,这是个重要的提醒。
还值得注意的是,他们中的许多人都是在可能被一些人看作远离科学研究的常规渠道的地方做出标志性工作的(西班牙的阿利坎特;法国的国防部;丹尼斯科的公司实验室;Vilnius在立陶宛)。此外,他们的重要论文都被一流期刊拒绝了——在很久的延迟之后得以发表在并不令人瞩目的位置。这些遭遇恐怕并非偶然:这些研究地点可能给予了从事不那么热门选题研究的更大自由,但是对于如何克服期刊和评审人的怀疑与不信任态度所给予的支持帮助却是较少的。
最后,这些科学突破背后的故事鲜有灵感迸发的瞬间。它们通常是一幕群戏,演出了10年以上,在其中的演职人员共同实现了他们各自孤身奋战所无法企及的高度。对于一般公众和希冀科学生涯的青年人,这都是精彩的一课。
原文链接:
https://doi.org/10.1016/j.cell.2015.12.041
来源:BioArt



1980-2020
原标题:《【学术前沿】特别致敬CRISPR幕后的英雄们——最全的一份CRISPR英雄谱》

