“人造生命体”合成酵母基因新进展:加装系统让菌株快速进化
由美国科学院院士Jef Boeke主导的国际科研项目——人工合成酵母基因Sc2.0,试图从酵母开始揭开“人工再造生命体”的神秘面纱,从“上帝”手中争夺“书写生命”的特权。
2017年3月10日,国际顶级期刊《科学》(Science)在同一天发表了上述项目的国际研究团队的7篇专刊论文。截至当时论文发表,16条酵母染色体合成了6条,其中彼时还是清华大学生命科学学院研究员的戴俊彪、天津大学化工学院教授元英进等率领的中国科研团队完成了工作量的66.7%。
一年之后,这支国际团队再次推出最新成果。北京时间5月23日凌晨,国际顶级期刊《自然》(Nature)子刊《自然•通讯》(Nature Communications)以专刊形式齐发7篇成果,中英美德四国科研团队围绕在人工合成酵母染色体上加装的SCRaMbLE 系统(Synthetic Chromosome Recombination and Modification by LoxP-Mediated Evolution)及其应用展开了一系列研究。
戴俊彪在接受澎湃新闻(www.thepaper.cn)采访时表示,“利用SCRaMbLE 系统,我们可以在短时间内快速地让合成的酵母染色体发生重新排列,这在传统基因组进化过程中是需要很多很多年的时间。”
戴俊彪目前是中国科学院深圳先进技术研究院合成生物学研究所合成基因组学研究中心的领头人,也是Sc2.0项目的主要负责人之一。他进一步解释,“现在我们只是针对一条染色体,未来如果整个基因组可以发生类似的基因重组,那两三天之内就可以发生翻天覆地的各种各样的重排。”
最新的研究进展还着重验证了SCRaMbLE 系统在工业应用菌株身上的潜力。实际上,对于工业上常用的酵母菌而言,SCRaMbLE 系统的“威力”在于可以迅速优化它们。或在短短几天时间内,大量的遗传变异体“池子”就可以被制造出来,研究人员可以按工业生产各取所需。
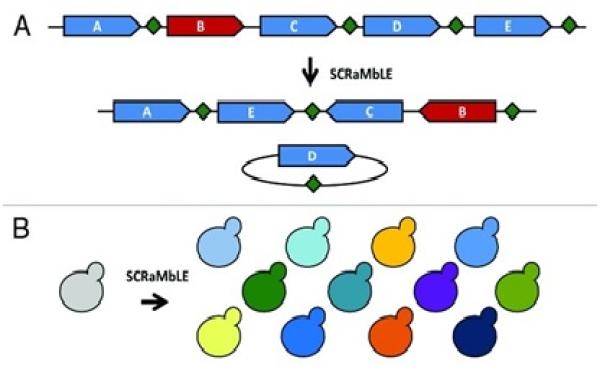
“加装”SCRaMbLE 系统
酵母菌株迅速进化的“超能力” 来自于SCRaMbLE 系统,然而SCRaMbLE 系统却不是天然存在的,是研究人员在人工合成酵母染色体时“加装”进去的。
“实际上,在设计合成染色体的时候,我们在每一个非必需基因的后面都掺入了一个LoxPsym位点,LoxPsym位点能被Cre重组酶识别。LoxPsym位点就相当于是特殊的标签,Cre重组酶进去之后抓出2个LoxPsym位点就能对对它们进行重组。“
回到Sc2.0项目,这一项目的目标是人工合成整个酵母的基因组。不过,Sc2.0项目目前仍保留了大量野生型基因组的DNA序列,同时只引入了少量新的改变。
其中一个改变就是特定重组位点的插入,即loxPsym位点,位于每个非必需基因终止密码子下游3bp(碱基)的位置。类似来源于P1噬菌体的天然LoxP位点,loxPsym位点能通过Cre重组酶促进直接重组。研究证实,SCRaMbLE可以介导长达几百Kb(千碱基对)范围的基因组重排。
戴俊彪解释,“合成酵母里面每条染色体上面可能都有几百个LoxPsym位点,Cre重组酶可以在不同的菌株里面随便抓2个进行重组,这就相当于整条染色体在大的菌株群体里面发生了各种不同的重排过程。”
毫无疑问,相比于传统繁殖、诱变,甚至现有的遗传工程等,SCRaMbLE系统下的这项新技可以在短时间内制造大量遗传变异体的“池子”。
戴俊彪表示,“想要诱导SCRaMbLE,第一合成染色体上要有很多不同的LoxPsym位点分布在不同的位置,第二要诱导表达Cre重组酶。而染色体重排以后会对这条染色体上的基因表达造成一定的影响,所以可以通过这点优化底盘细胞,也就是让细胞对某一种性状更适应。”
值得一提的是,SCRaMbLE系统早在2011年就首次发表于《自然》,由Boeke率领的团队完成研究。在当时的研究中,携带部分人工合成染色体(插入43个LoxPsym位点)的菌株在重组酶激活后最终产生了突变菌株,这些突变株在生长率方面表现出了很大的变化。同时还证明了重组仅仅发生在LoxPsym位点。
戴俊彪称,研究人员将这一系统命名为SCRaMbLE系统,暗示着原基因组被彻底“打乱了”(scramble)。
戴俊彪和英国曼彻斯特大学蔡毅之的合作队伍还为SCRaMbLE系统设计了一套筛选系统ReSCuES,这套系统被形象地称为神奇的“进化加速器”。
在细胞内的野生型loxP位点和loxPsym位点具有严格的正交性,即不会发生交叉反应,但是都能够被Cre重组酶识别并介导反应。利用这一特性,研究团队构建了一个正交性的报告系统ReSCuES,可以从SCRaMbLE后的混乱群体中精确筛选出发生基因组重排的细胞。
此外,英国帝国理工学院Tom Ellis团队还重点对携带了一条完全人工合成染色体(5号染色体)的酵母进行了应用研究。用SCRaMbLE系统改善药物合成,并使菌株适应另一种替代糖原的代谢。
研究团队将盘尼西林生物合成途径放入到酵母中,通过SCRaMbLE系统对合成染色体进行基因重排,最终盘尼西林产量提高了2倍。他们还用SCRaMbLE系统来改进酵母菌株,提高酵母对替代糖原木糖的利用。
如何避免“重组”死亡
不过,这样彻底“打乱”后爆发式般的重组带来的菌株快速进化方式略显粗暴。
《自然•通讯》此番就酵母合成染色体重排成果还同时发表了一篇评论,评论中即指出,删除或增强某些基因可能是致命的。戴俊彪也提到这一问题,“如果是单倍体酵母的话,一旦把重要的基因去掉有可能就不能活了。”
在最新的研究中,就SCRaMbLE系统带来的大量重组或将引发的致死问题也进行了改进设计。
Boeke和他的同事试图解决这一问题 ,他们将合成染色体和野生型染色体混合。戴俊彪介绍,“之前所有的文章在做基因重排的时候,都是基于酿酒酵母的单倍体菌株,所谓的单倍体就是它只有一套染色体,从1号到16号。”
Boeke等人在一条是合成型、另一条是野生型的异源二倍体菌株里面去诱导SCRaMbLE重排。“这个好处就是因为有野生型染色体的存在,即使另一条合成染色体发生了很大的重组,很多细胞还是可以存活的。”
研究证明,二倍体的“后代”比单倍体菌株生命力更“顽强”。例如,在42摄氏度的条件下,酵母仍然能生长,在高咖啡因水平暴露中也能继续存活。
人类基因组合成还有多远
据戴俊彪介绍, Sc2.0项目需要完成的16条酵母染色体的合成目前完成了90%左右。“我们希望到2018年年底把所有的片段都能合成完。”
然而,Boeke等人更宏伟的另一项计划、早在2016年6月就宣布启动的 “人类基因组编写计划”( GP-write)进展又如何?这一计划意味着“人造生命体”最终触及人类。
据《科学》官网5月1日报道,需要筹资1亿美元的GP-write项目因目标过于宏大受到争议而得不到任何资金支持。Boeke等发起者们迫于压力不得不优先提出一个“缩减版”项目:合成防病毒感染的人类细胞。
戴俊彪积极推动的“国际基因组编写计划•中国”( GP-write China)也已于2017年12月2日在深圳正式启动。
针对GP-write项目的可行性,戴俊彪表示,“我觉得GP-write很重要,但是现在很难拿出一个很现实的方案去执行。”在戴俊彪看来,合成整个人类基因组不仅仅是单纯为了合成而合成,“这只是证明合成和原来一样的基因组的能力。”
既然如此,下一个问题就是引入设计。“意味着你要放入一个什么样的设计在里面?这是整个GP-write项目一直在探讨的,你希望人类基因组里面的设计原则是什么,你希望改变什么?”
除了设计之外,技术是GP-write项目的另一项挑战。“我们现在的技术水平去实现30亿个碱基对是很难的,甚至包括把一个大的片段DNA放进一个人类细胞里面去也是很困难的。”戴俊彪提到。
当然资金问题也的确存在。戴俊彪表示,“虽然合成成本在不断下降,但乘上30亿以后也是非常大的数字。我们能不能发展出一套新的合成技术,把基因合成的成本降2个数量级甚至3个数量级?那我们就可以真正去做这些事情了。”目前,在大量合成情况下,合成成本基本在每个碱基需要5美分-8美分。
戴俊彪强调,“整个GP-write是一个大的目标,我们希望借助这样一个大的目标去推动其他事物,比如提高设计、提高技术,这才是它最大的贡献。”
至于GP-write China,戴俊彪提到,“我们现在正在确定到底该怎么样去做,是不是直接去做人类的,还是去做其他生物的。我们希望有一个适合中国这样一个大研究背景下的基因组设计。”
就目前研究的酵母染色体合成工作而言,戴俊彪表示,“后续我们实验室希望能对酿酒酵母的基因组有一个更深层次的设计,现在Sc2.0做的研究仅仅是基于野生型做了一些小的修改”戴俊彪提到,Sc2.0目前对整个基因组的结构、基因排列的顺序、基因的位置,以及基因的调控元件等并没有做任何变化。
