以下文章来源于BioArt ,作者小白薯

高屋建瓴,提供专家点评,引导学术争论,展现学术批评;诚心实意,关注科研生态,推广科研经验,倡导师生交流。

获取更多CSCB2020相关资讯
编者按
spliceosome(剪接体)和nuclear pore complex(核孔复合物)结构曾被Nature杂志评为结构生物学领域两个最被期待解决的重大科学问题(亦被称为两大“圣杯”)。施一公教授在国内带领的研究团队经过数年的努力,目前已经基本完成spliceosome拼图()。然而意想不到的是,施老师团队近日两篇在预印本上发布的高分辨率核孔复合物结构再一次震撼了结构生物学领域,可能因为疫情的原因,没有太多人关注这个重要的超大复合物(哺乳动物细胞的核孔复合物相对分子质量高达1亿道尔顿)。2018年,Nature杂志罕见发表了一篇有5位重量级通讯作者(分别是洛克菲勒大学Michael P. Rout和Brian T. Chait、UCSF的Andrej Sali、波斯顿大学Christopher W. Akey和贝勒医学院的Steven J. Ludtke )的核孔复合物结构论文,报道了酵母Yeast细胞核孔复合物低分辨率冷冻电镜结构(),该结构包括552个蛋白组分,整体分辨率为28Å ,内环结构20-25Å,当时被认为是最完整的核孔复合物。当时,BioArt在报道相关工作的时候曾预言,“在未来两到三年甚至更短的时间里,核孔复合物将会被解析到近原子或原子分辨率,从技术上来说不是问题。并且我国科学家也很有可能在这场“竞赛”中取得优异成绩”,没想到近原子分辨率的核孔复合物结构(胞质环)这么快就到来。
本次,两篇施老师团队的预印本论文中解析的解析了来自非洲爪蟾NPC的胞质环的近原子分辨率结构以及环绕NPC的腔环结构。祝贺施老师团队!相信在不久的将来,NPC的原子拼图也将由施老师团队等率先完成。
这么好的工作,CNS可能会发邮件直接约文章了!极大概率两篇背靠背Science。
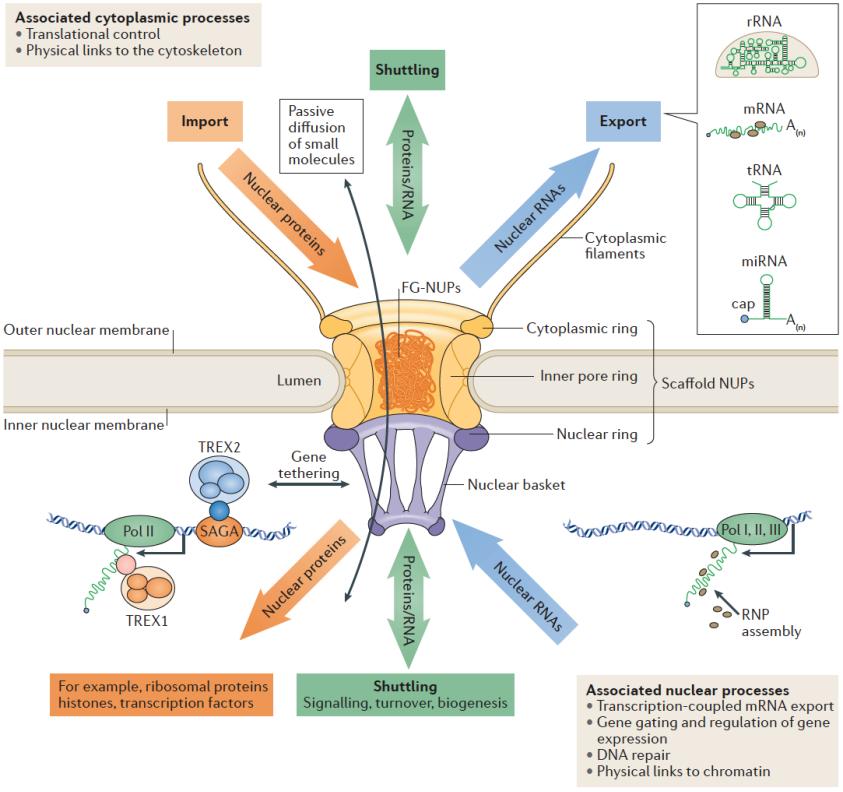
真核生物中,最重要的遗传物质DNA位于细胞核内,而最重要的功能载体—蛋白质—其合成及发挥功能却是在细胞核外或者需要进出细胞核发挥功能。因此需要一个通道允许这些生物分子进出细胞核,而组成这个通道的生物大分子就是核孔复合物(Nuclear Pore Complex, NPC)。NPC是真核生物连接胞质和细胞核的唯一双向通道,它控制着所有的生物大分子进出细胞核,功能重要性不言而喻。它分子量巨大,是细胞结构中分子量最大的生物大分子。在酵母中,NPC分子量约为50 MDa;在高等真核生物中,其分子量达到110-125 MDa。因此,NPC是最重要的分子机器之一【1】。
要想知道蛋白和和核酸是如何进出细胞的分子机制,前提就得了解NPC的运作机制,而结构的解析是这一切的先决条件。
3月27日,预印本网站bioRxiv更新了两篇来自施一公课题组关于最新NPC精细结构的研究论文。其中一篇题为“Structure of the Cytoplasmic Ring of the Xenopus laevis Nuclear Pore Complex”,该篇论文介绍的是—通过冷冻电镜技术(cryo-electron microscopy, Cryo-EM),解析了来自非洲爪蟾NPC的胞质环(cytoplasmic ring, CR)的近原子分辨率结构,平均分辨率5.5-7.9 Å,局部分辨率4.5 Å。另一篇文章(清华大学李赛为共同通讯作者)题为“Molecular architecture of the luminal ring of the Xenopus laevis nuclear pore complex”,主要通过冷冻电镜断层成像技术(cryo-electron tomography, Cryo-ET), 解析了整个NPC复合物及环绕NPC的腔环(Luminal ring, LR)的原位结构,其中NPC复合物的胞质环 CR, 内环(inner ring, IR), 核环(nuclear ring, NR)的分辨率分别为9.1 Å, 13.1 Å, 13.6 Å, LR的分辨率为15.1 Å。

NPC的结构解析工作开始于大约20多年前【3】,目前已经有通过X-ray解析的含单个或部分组分的晶体结构;以及通过Cryo-EM和Cryo-ET解析的低分辨率复合物结构。在此前,关于NPC的CR最高分辨率为15 Å;通过Cryo-ET解析了来自五个不同物种的原位结构,分别是盘基网柄菌(D. discoideum)【4】,酿酒酵母(S. cerevisiae) 【5】,莱茵衣藻(Chlamydomonas reinhardtii)【6】,非洲爪蟾(X. laevis) 【7】和人(H. sapiens)【8】,分辨率分别为58 Å, 28 Å, 30 Å, 20 Å, 和23 Å;而关于LR的整体结构还是个迷。
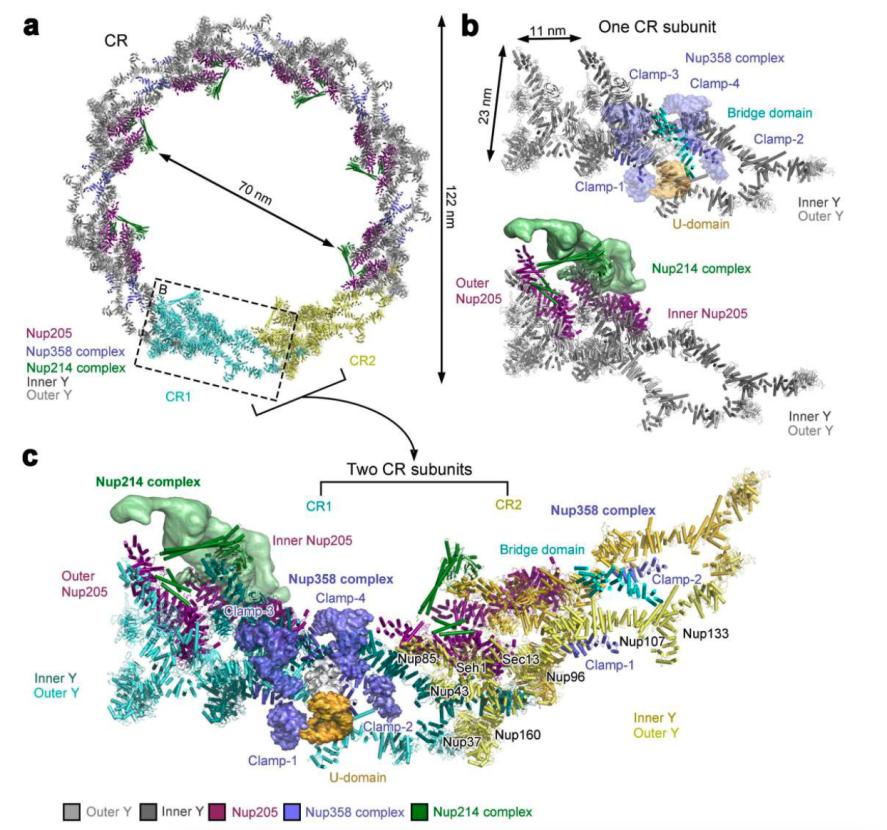
在施老师的文章中,他们首先通过Cryo-EM解析了来自非洲爪蟾的CR的近原子分辨率结构。最后的结构模型核心区域平均分辨率为5.5 Å,包含Nup358区域7.1 Å,包含Nup214区域7.9 Å,局部分辨率达4.5 Å (图2)。从结构中可以看到,CR呈现为一个旋转对称的圆形结构,由八个重复CR单元组成。CR环的外环和内环直径分别为122 nm和70 nm。
该复合物结构达到如此高分辨率尚属首次,相比于此前通过Cryo-EM和Cryo-ET的解析结果,该结构模型得到了巨大的改善。在该分辨率下,可以相对更加清晰准确地将基本的蛋白二级结构和大部分的CR组分修入模型当中。该结构为此后原子分辨率的模型搭建提供了指导方向。
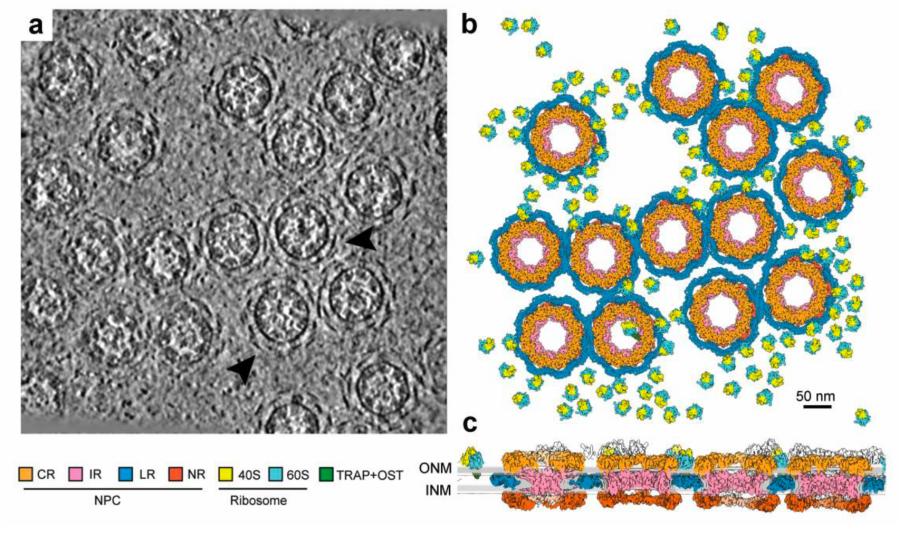
在施老师的另一篇文章中,他们通过Cryo-ET和Cryo-EM技术相结合,重构了NPC复合物的原位结构;其中LR的核心特征结构是完全不依赖于Cryo-EM技术实现的(图3)。在样品处理的过程中,CR, IR, NR, 和LR的单颗粒分别收集和处理。通过重构,可以分别得到9.1 Å, 13.1 Å, 13.6 Å, 和 15.1 Å的平均分辨率。为了更加真实的显示是原位的样品,他们还收集了780个核糖体颗粒进行三维重构,最终得到的分别率为16.4 Å(图3)。
通过Cryo-ET技术,可以直接的观察到所有的生物大分子在非洲爪蟾NE的原始断层结构。基于结构模型的相对位置,作者在原始断层位置建模重构了CR, IR, NR, LR和核糖体的亚基单元。这些断层重构模型揭示了CR, IR, NR, LR和核糖体在体内真实状态的空间组成和位置。从模型中可以看到,几乎每一个NPC都会被其他NPC或核糖体围绕;同时,LR会环绕NPC并且填满INM和ONM之间的空间(图3)。
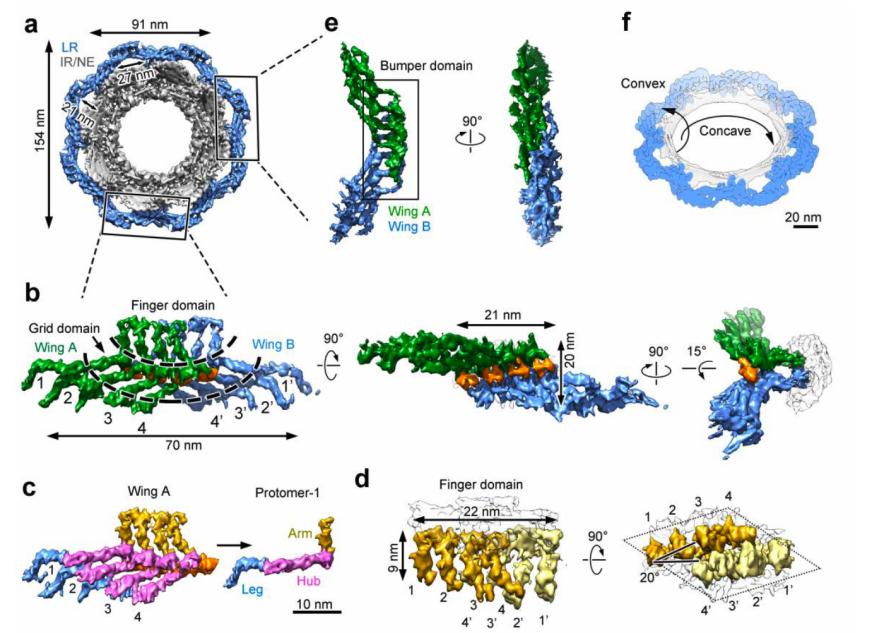
所有的这些通过Cryo-ET重构(sub-tomogram averaging, STA)的结构模型都单独的进行了Cryo-EM重构(single particle analysis, SPA)过程,结果显示这些结构可以很好的在空间上比对在一起,表明Cryo-ET重构的模型至少在技术上是可重复的(图4)。整体NPC结构内环直径49 nm, 没有LR的外环直径122 nm (与CR一致),带有LR的外环直径为154 nm (图4)。
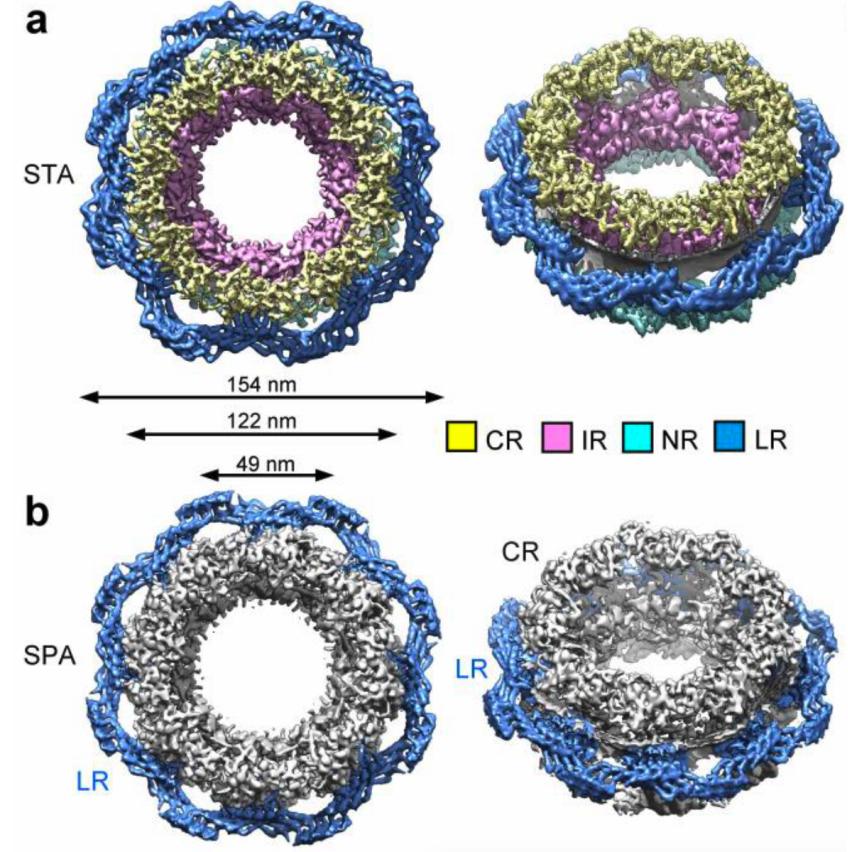
LR结构包含八个蝴蝶状重复单元,每一个单元包含两个对称的“翅膀”。每一个翅膀由四个延长的管状原体组成。在LR的单个单元内,这八个原体形成了两个结构域,其中一个为Finger结构域,该结构与直接接触在INM和ONM间的融合处;另一个为Grid结构域,它作为Finger结构域的坚实基础(图4,5)。两个邻近的LR单元间,通过“翅膀”的侧面边缘与对方相互作用,并由此形成了一个Bumper结构域,似乎起着缓冲邻近NPC的作用 (图5)。该结构揭示了许多此前对于LR的不清楚的特征,并且可以为NPC的弹性性质提供一定的解释。
这两篇文章为核孔复合物领域注入了新鲜的血液,这次分辨率的提高是该领域的一个重大进展,它即将开启原子分辨率的大门,让我们有幸能够看到核孔更加精细的原子结构,了解相关的分子机制。
更让我们惊讶和狂喜的是,曾被《Nature》杂志誉为结构生物学领域两个最被期待解决的重大科学问题—spliceosome和nuclear pore complex【9】,在施老师完成大部分spliceosome拼图后,又成功进军NPC领域。相信在不久的将来,NPC的原子拼图也将完成。
原文链接:
https://www.biorxiv.org/content/10.1101/2020.03.27.009407v1
https://www.biorxiv.org/content/10.1101/2020.03.27.009381v1
参考文献
1. Beck, M. et al. Nature reviews Molecular cell biology 18, 73, (2017).
2. Akey, C. W. et al. The Journal of Cell Biology 122, 1-19, (1993).
3. Hinshaw, J. E. et al. Cell 69, 1133-1141, (1992).
4. Beck, M. et al. Nature 449, 611-615, (2007).
5. Kim, S. J. et al. Nature 555, 475-482, (2018).
6. Mosalaganti, S. et al. Nature communications 9, 1-8, (2018).
7. Eibauer, M. et al. Nature communications 6, 7532, (2015).
8. von Appen, A. et al. Nature 526, 140-143, (2015).
9. Jones, N. Nature 505, 602-604, (2014).
来源:BioArt


原标题:《【学术前沿】施一公团队解析核孔复合物近原子分辨率胞质环结构及高分辨率原位核孔复合物腔环结构》
