原创 Cell Press CellPress细胞科学

交叉学科
Interdisciplinary
深度学习是一套强大的神经网络学习技术,目前为许多问题提供了最佳解决方案,包括从抗体的氨基酸序列和B细胞受体库中识别未检测到的模式。
本期精选的两篇文章在抗体研究中使用深度学习方法来理解公共抗体应答(Immunity的文章),以及提出关于抗体功能和结合位点预测的见解,以帮助发现新的治疗方法并揭示疾病的生物学(Patterns的文章)。这些文章强调了计算方法的影响,及其在免疫学中的应用。
使用自监督学习破译抗体的语言
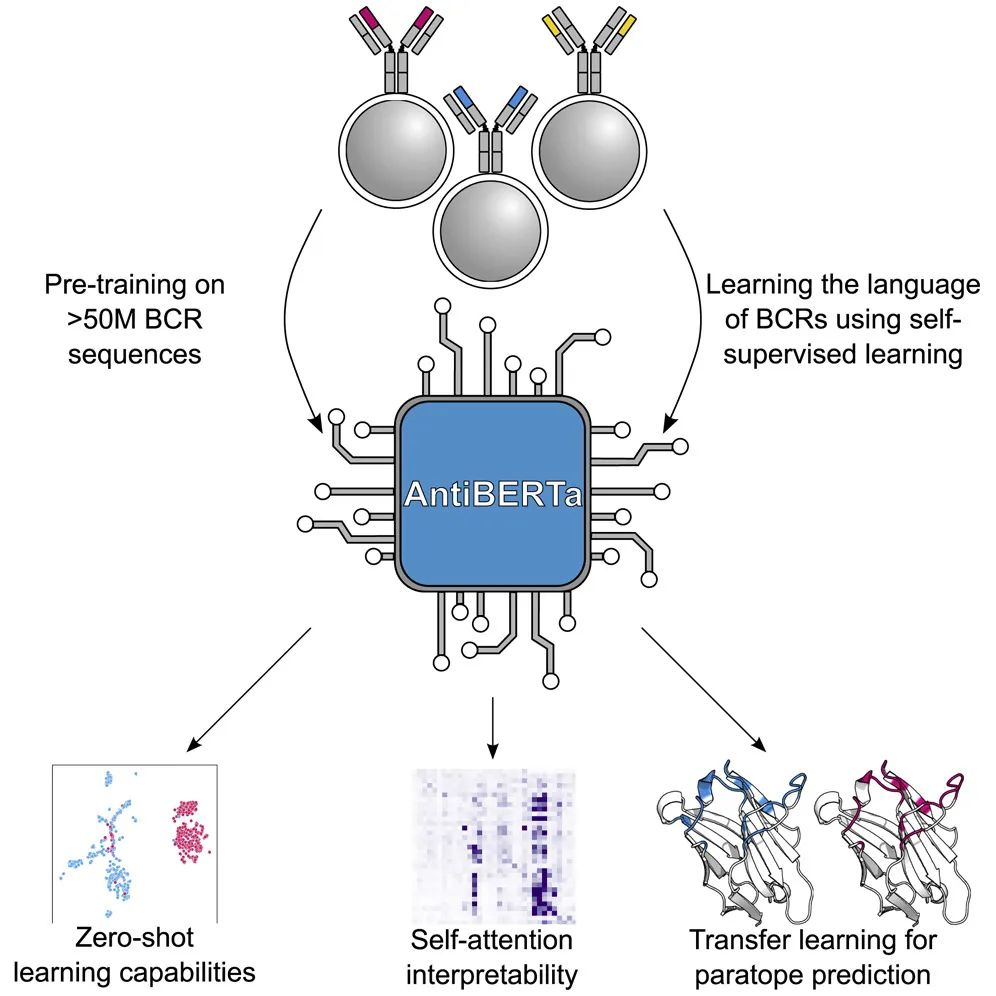
了解抗体功能对于解读疾病的生物学以及发现新的治疗性抗体至关重要。但面临的挑战是,目前可用的标记数据很有限,但抗体变体存在巨大的多样性。来自英国的抗体治疗公司 Alchemab Therapeutics的Jinwoo Leem团队在Patterns发表研究论文,为了克服上述挑战,他们使用自我监督学习来训练一个大型抗体特异语言模型,然后使用迁移学习来微调模型,以预测与抗体功能相关的信息。研究团队提供了抗体结合点预测的领先结果,从而初步证明该模型是成功的。这一模型可针对不同应用进一步微调,以提高对抗体功能的理解。
团队负责人Jinwoo Leem表示,其研究工作强调了深层语言模型在理解抗体结构和功能方面的巨大潜力。他们开发的模型AntiBERTa是一个转换神经网络,可以读取抗体氨基酸序列的组成部分,揭示诸如突变负荷和抗体结构等模式。另外,作者在文中展示了AntiBERTa如何专门用于与抗体药物发现有关的许多不同应用,如互补位或结合点预测。
一项大规模系统调查揭示了针对SARS-CoV-2的公共抗体应答反复出现的分子特征
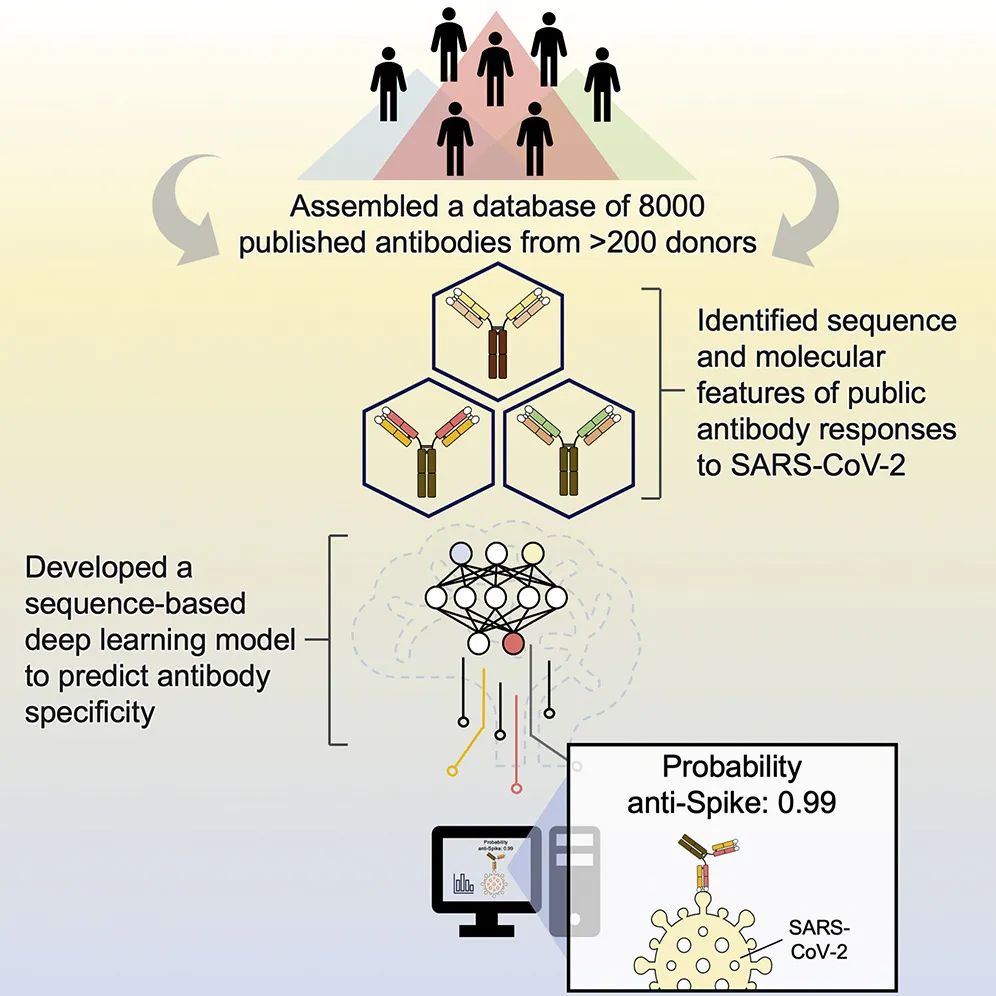
在抗击COVID-19大流行的全球研究中,已经分离并表征了数千种针对SARS-CoV-2突起蛋白的人类抗体,为研究针对单一抗原的抗体反应提供了前所未有的机会。来自伊利诺伊大学厄巴纳-香槟分校的Nicholas C. Wu团队在Immunity发表研究论文,利用从88篇研究发表和13项专利中获得的信息,收集了来自超过200名捐献者的约8000种针对SARS-CoV-2突起蛋白的人类抗体数据集。通过分析免疫球蛋白V和D基因的使用、互补决定区H3序列和体细胞高频突变,作者证明了对突起蛋白不同结构域的共同(公共)反应是完全不同的。作者进一步使用这些序列来训练深度学习模型,以准确区分针对SARS-CoV-2突起蛋白的人类抗体和针对流感血凝素蛋白的抗体。总的来说,这一研究为抗体研究提供了信息资源,增强了我们对公共抗体应答的分子理解。
团队负责人Nicholas C. Wu表示,关于SARS-CoV-2单克隆抗体的已公开或发表资料的数量巨大,并且还在继续增长。其团队的研究表明,深入挖掘这些信息可以为针对SARS-CoV-2的抗体反应提供先前未知但十分重要的见解。
